
2024.6
Collaboration with
 との連携
との連携
創薬、構造生物学コミュニティにおけるFMOデータの利用拡大に向け、2023年度から
In order to expand the use of FMO data in the drug discovery and structural biology communities, we have started a collaboration between
 (https://pdbj.org) と
(https://pdbj.org) and
(https://pdbj.org) と
(https://pdbj.org) and

(https://drugdesign.riken.jp/
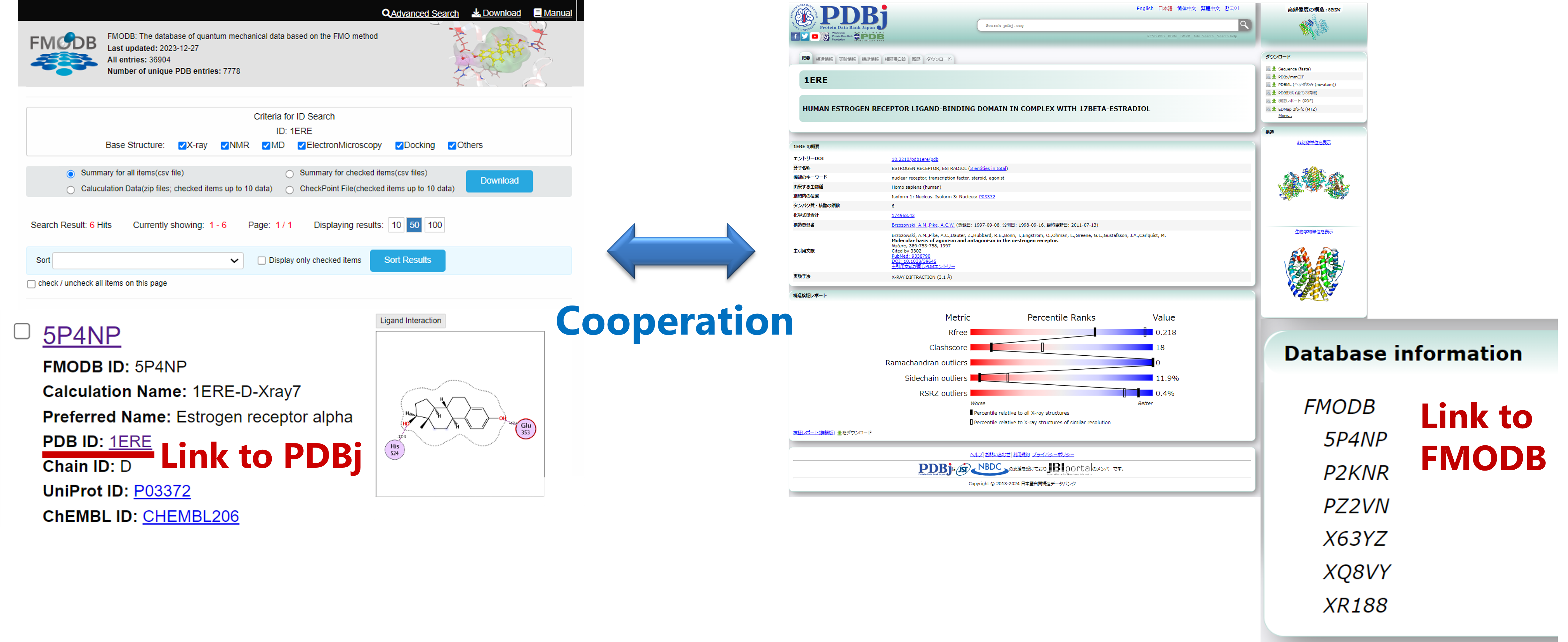
データ活用における利便性向上の第一歩として、HP上の各PDB、FMODBデータに対して、それぞれ関連するFMODB ID, PDB IDの相互リンクを配置することで、互いのデータに簡便なデータアクセスが可能となりました(図1)。
FMODBのデータセットは、年度ごとに確実に蓄積し、X線結晶構造だけでなく、PDBに登録される他の実験構造、MDスナップショットデータなどの分子シミュレーション構造のデータ蓄積も進んでいます。
エントリー数の詳細な数値は表1、元構造の種類についての解析は図3に詳細を示しました。
(https://drugdesign.riken.jp/FMODB/) in FY2023, in cooperation with Prof. Genji Kurisu and Dr. Gert-Jan Bekker at Institute for Protein Research, Osaka University.
As a first step to improve data utilization convenience, we have placed mutual links by the related FMODB ID and PDB ID for each PDB and FMODB data on the HP to allow easy access to each other's data (Figure 1).
The FMODB dataset has been steadily accumulating year by year, including not only X-ray crystal structures but also other experimental structures registered in the PDB and molecular simulation structures such as MD snapshot data (Table 1, Figure 3).
Table 1 shows detailed figures on the number of entries, and Figure 3 detail analyses of the types of original structures.
Cooperation with PDBj: Improvement of convenience by mutual linkage (Figure 1)
Growth of FMODB data per year (Figure 2)
Composition of the original structure of FMODB (Figure 3, Overall Data Distribution (a) and Detailed Breakdown of 'Others' (b)))
Number of each data on FMODB (Table 1)
Acknowledgments
本データベース開発者は、大阪大学蛋白質研究所の栗栖 源嗣 教授とGert-Jan Bekker 博士に、PDBjとFMODBの連携に関して有益な議論と技術的支援を頂けたこと感謝致します。
The developers of FMODB would like to thank Prof. Genji Kurisu and Dr. Gert-Jan Bekker of Institute for Protein Research, Osaka University for fruitful discussions and technical support of the collaboration with PDBj.